L’utilisation des très hauts champs magnétiques améliore sensiblement la résolution spatiale des données d’imagerie par résonance magnétique (IRM) sans toutefois atteindre l’échelle microscopique qui permet de visualiser les cellules individuelles. Des chercheurs du CEA-Joliot (NeuroSpin) et leurs collaborateurs du Centre de recherche de Juelich[1] développent une approche originale qui repose sur la simulation numérique à large échelle pour entraîner des algorithmes d'apprentissage automatique (machine learning) à décoder in vivo l’organisation cellulaire des tissus cérébraux de volontaires sains ou de patients à partir de simples acquisitions d’IRM de diffusion, sensibles au mouvement microscopique de l’eau à l’intérieur des tissus. Cet outil, fondé sur l’utilisation conjointe de simulations du signal IRM et de méthodes d’intelligence artificielle (IA), vise à répondre au besoin de la recherche fondamentale sur le cerveau humain, en particulier du décodage in vivo de la cytoarchitecture du cortex cérébral, mais aussi au besoin de la recherche clinique afin de mettre à disposition des cliniciens un substitut à la biopsie qui reste un acte chirurgical très invasif.
L’outil de décodage repose sur trois étapes de simulation nécessitant l’utilisation d’infrastructures de calcul à haute performance (HPC). Il faut générer une collection d’échantillons virtuels ultra-réalistes du tissu cérébral, et pour chacun d’eux simuler la diffusion de l’eau puis le signal d’IRM de diffusion qui serait obtenu.
L’équipe détaille le principe du nouvel algorithme MEDUSA (pour Microstructure Environment Designer with Unified Sphere Atoms) développé pour simuler les échantillons virtuels ultra-réalistes. Les chercheurs démontrent que MEDUSA permet de synthétiser la géométrie des membranes de populations de cellules correspondant à n’importe quelle région cérébrale. Reposant sur la prescription d’un jeu de quelques dizaines de paramètres, l’algorithme permet de créer des échantillons virtuels de tissu avec un degré de réalisme encore jamais atteint in silico.
Ce nouvel outil permet désormais de simuler le signal IRM attendu pour toute configuration de populations de cellules à même de peupler le tissu cérébral, et donc d’apprendre la signature IRM propre à chacune de ces configurations en entraînant une méthode de machine learning. Une fois entraînée, la méthode peut alors être utilisée pour décoder l’organisation cellulaire à partir d’un jeu réduit de signaux IRM, constituant ainsi un véritable outil permettant de réaliser des biopsies virtuelles.
L’outil développé fait d’ores-et-déjà l’objet de premières campagnes de simulation au sein du Très Grand Centre de Calcul du CEA (CCRT, Bruyères-le-Châtel), afin de démontrer qu’il est possible de quantifier précisément le gonflement des axones qui survient lors d’un accident vasculaire cérébral. Une campagne de plus grande ampleur pour simuler une dizaine de milliards d’échantillons virtuels fait actuellement l’objet d’une demande de ressources auprès du GENCI[2] afin de permettre la cartographie in vivo de la microstructure de la substance blanche cérébrale.
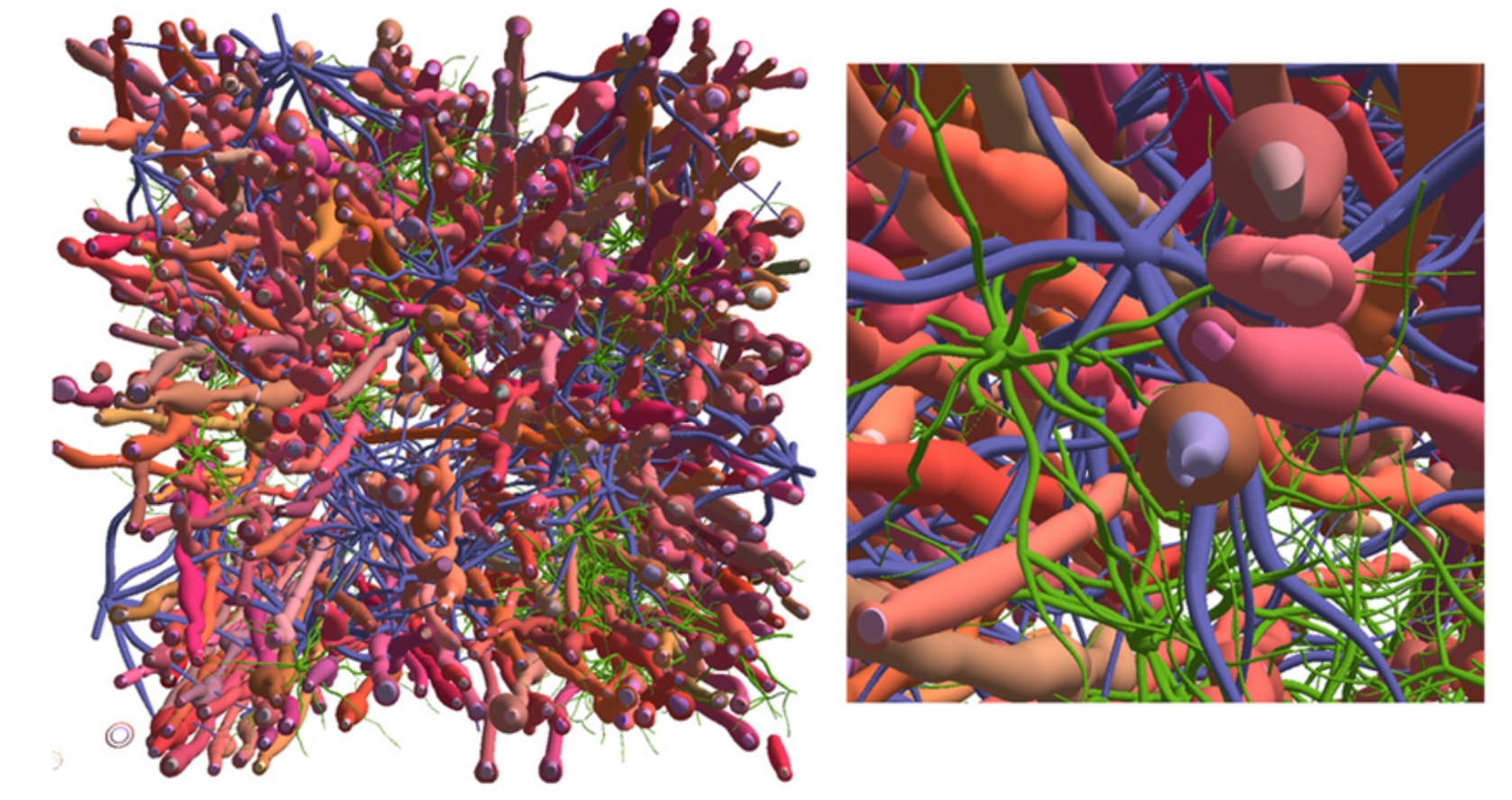
Synthèse d’un échantillon cubique de 100 micromètres de côté, représentatif de la substance blanche cérébrale, et généré à partir du simulateur MEDUSA, incluant près de 200 axones myélinisés, ainsi que de nombreux astrocytes et oligodendrocytes. © K. Ginsburger / C. Poupon / CEA
[1]Institute of Neuroscience and Medicine INM, Juelich, Allemagne
[2] Grand équipement national de calcul intensif