Si le Vivant est composé essentiellement de matière organique, une très grande quantité de processus naturels dépend directement de facteurs inorganiques. Ainsi, près de 40 % des protéines ne fonctionnent que parce qu’elles fixent un ou plusieurs ions métalliques (sodium, magnésium, calcium, fer, zinc, cuivre, etc.). Ce sont les métalloprotéines. Ces protéines contiennent ainsi des agrégats inorganiques qui interviennent dans des réactions biosynthétiques et métaboliques d’une très grande importance pour la vie cellulaire. Par exemple, les agrégats fer-soufre [Fe-S], ubiquitaires chez les animaux, les plantes et les bactéries sont essentiels pour la catalyse enzymatique, le transfert d’électrons (respiration, photosynthèse) ou la régulation de l’expression des gènes.
Des chercheurs du CEA-Irig (IBS) se sont intéressés à la métalloprotéine bactérienne RsrR comportant un centre [2Fe-2S]. RsrR participe à la régulation métabolique de la cellule, en réprimant l’expression de certains gènes. Début 2019, les scientifiques ont déterminé sa structure cristalline grâce à des études de diffraction aux rayons X et leurs collaborateurs britanniques ont montré que la fixation à l’ADN ne se fait pas lorsque la protéine est réduite, c’est-à-dire lorsque son centre [2Fe-2S] capte un électron. Récemment, ils sont allés plus loin dans l’explication du phénomène.
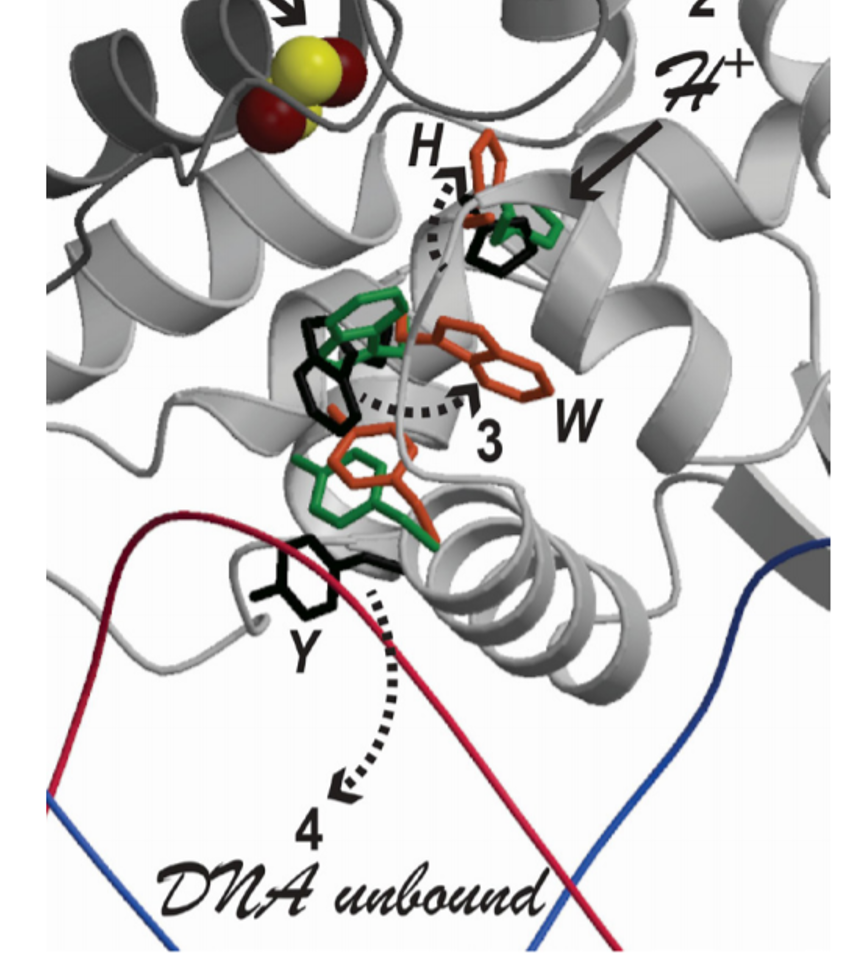
Leur hypothèse ? « Nous avons supposé, grâce aux structures cristallines, que lorsque la protéine est réduite, la chaîne latérale d’un résidu d’acide aminé tryptophane se tourne vers l’intérieur de la protéine ce qui empêche sa fixation à l’ADN », explique Juan C. Fontecilla-Camps, chercheur à l’Irig (IBS). Pour vérifier cela, les scientifiques ont tenté de modifier chimiquement le tryptophane dans les deux configurations, réduite (avec un électron - de charge négative - en plus), ou oxydée (sans cet électron). Dans le premier cas, ils n’y sont pas parvenus, validant l’hypothèse de l’enfouissement du tryptophane à l’intérieur de la protéine RsrR (il n’est alors plus accessible). En revanche, le tryptophane a pu être modifié dans la configuration oxydée, confirmant le fait qu’il est exposé au solvant.
Les chercheurs ont également levé le voile sur le comportement d’un autre acide aminé, une histidine, concomitant à celui du tryptophane. « Lorsque RsrR est réduite, cette histidine se déplace vers le centre métallique de la protéine », ajoute Juan C. Fontecilla-Camps. « Nous avons montré par des calculs théoriques (collaboration CEA-Irig (DIESE)) que la chaîne latérale de cet acide aminé est alors protonée et que, grâce à sa charge positive, elle est attirée par le centre [2Fe-2S]. Cette histidine laisse alors la place au tryptophane pour s’enfouir dans la protéine. » Finalement, cette hypothèse a été confirmée par la structure cristalline d’un complexe RsrR-ADN.
Ces travaux montrent comment des effecteurs aussi petits qu’un électron et un proton vont pouvoir induire des changements structuraux responsables d’une réponse adaptée de la bactérie à son environnement !
(c)IBS