Une bactérie est dite à Gram positif ou à Gram négatif suivant sa réaction à un test de coloration appelé test de Gram. C’est le premier test à être effectué lorsque l’on cherche à identifier la bactérie responsable d’une infection lors d’un diagnostic bactérien.
Nous avons développé en collaboration avec l’EPFL (Suisse) un dispositif de piégeage optique basé sur l’utilisation de cristaux photoniques 2D à cavités creuses et dédié à l’étude des bactéries. Le piégeage de sept types de bactéries a été démontré sur ces structures optiques :
Pseudomonas putida,
Neisseria sicca,
Escherichia coli,
Yersinia ruckeri,
Bacillus subtilis,
Listeria innocua et
Staphylococcus epidermidis.
Le signal de piégeage de ces bactéries transmis par la structure optique est caractérisé par deux phénomènes : une brusque variation du niveau d’intensité transmise et de fortes oscillations liées au déplacement de la bactérie lorsqu’elle est en interaction avec le champ de la cavité. Nous montrons que la combinaison de ces deux caractéristiques permet l’identification du type de Gram des bactéries étudiées et parfois leur identification absolue parmi cet échantillon de sept espèces bactériennes.
Cette technique permet donc l’identification du type de Gram des bactéries par une méthode rapide, ne nécessitant pas d’ajout d’agents chimiques et permettant de travailler à faible concentration de bactéries. De plus cette méthode est non-destructive, contrairement au test de Gram classique, et permettrait donc de réaliser d’autres tests sur une même bactérie, favorisant leur identification.
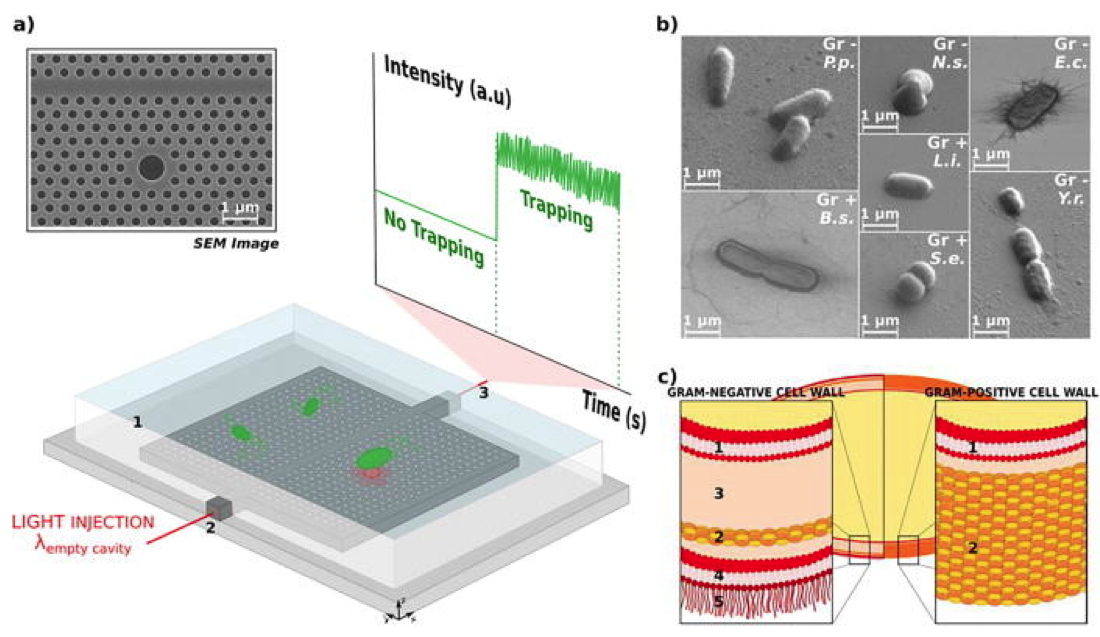
Figure : (a) Image MEB de cristaux photoniques 2D à cavités creuses et montage expérimentale. Les structures à cristaux photoniques sont immergées dans l'eau (1) grâce à un bassin PDMS qui autorise le mouvement brownien des bactéries à proximité de la cavité optique. La lumière d'un laser accordable est injectée à la longueur d'onde de résonance dans le guide d'ondes (2). La lumière transmise est collectée à partir de l'autre facette de l'échantillon (3)
via un objectif de microscope et son intensité est contrôlée à l'aide d'un oscilloscope. (b) images MEB des sept bactéries étudiées. P.p. signifie
Pseudomonas putida, N.s. pour
Neisseria sicca, E.c. pour
Escherichia coli, Y.r. pour
Yersinia ruckeri, B.s. pour
Bacillus subtilis, L.i. pour
Listeria innocua et S.e. pour
Staphylococcus epidermidis. Le type de Gram de ces bactéries est également indiqué : Gr - pour les bactéries Gram-négatives et Gr + pour les bactéries Gram-positives. (c) Description des différences structurelles dans la paroi cellulaire pour les bactéries à Gram négatif et à Gram positif. Les bactéries Gram - présentent deux membranes (plasma 1 et externe 4) séparées par un espace périplasmique liquide (3) et par une couche mince de peptidoglycane (5 à 10 nm) (2). De plus, les lipopolysaccharides (LPS, 5) font saillie de la membrane externe. Au contraire, les bactéries Gram + présentent une structure moins complexe, avec une seule membrane (plasma 1), entourée d’une enveloppe de cellules épaisses (20–80 nm) constituée d’une couche de peptidoglycane (2) et d’aucun LPS.