Les mécanismes centraux affectant la vie des bactéries sont contrôlés par des réseaux de régulation complexes permettant une adaptation à un environnement en constante évolution. Le genre bactérien
Pseudomonas comprend plus d’une centaine d’espèces aux capacités d’adaptation exceptionnelles. Largement répandues dans l’environnement, elles sont présentes dans de nombreux écosystèmes différents, tels que le sol, les eaux, les plantes, les animaux et même les nuages. Cette adaptation implique généralement la reprogrammation de l'expression de gènes nouvellement acquis afin d'ajuster leur expression aux besoins spécifiques de l’environnement rencontrés par la bactérie receveuse.
Dans de précédentes études, des chercheurs de l’Irig ont identifié la protéine Exolysine A comme étant un facteur de virulence majeur de différentes espèces de
Pseudomonas. Cette toxine provoque la mort cellulaire grâce à la formation de pores dans les membranes de son hôte. Elle est codée par un
opéron (opéron
exlBA) acquis par
transfert horizontal de gènes. Du fait de la diversité d’environnements de ces bactéries, ces chercheurs, en collaboration avec une équipe de Harvard Medical School (Boston, USA), se sont intéressés à la régulation de la synthèse de cette toxine dans plusieurs espèces de
Pseudomonas.
En étudiant différentes souches cliniques de
P. aeruginosa, ils ont découvert l’existence de ErfA, un inhibiteur impliqué dans l’expression de l’opéron
exlBA. L’étude du
régulon d’ErfA montre que sa cible principale est sans rapport avec la toxicité liée à l’Exolysine A et que la régulation de l’opéron
exlBA, donc de la virulence, est spécifique à l’espèce
P. aeruginosa.
Les chercheurs ont ensuite étudié les
régions régulatrices de l’opéronexlBA de 446 souches de
Pseudomonas (Figure). Cette analyse leur a permis de montrer que certains éléments de régulation sont apparus spécifiquement chez
P. aeruginosa, alors que les autres espèces de
Pseudomonas possèdent des motifs prédits pour être reconnus par d’autres régulateurs.
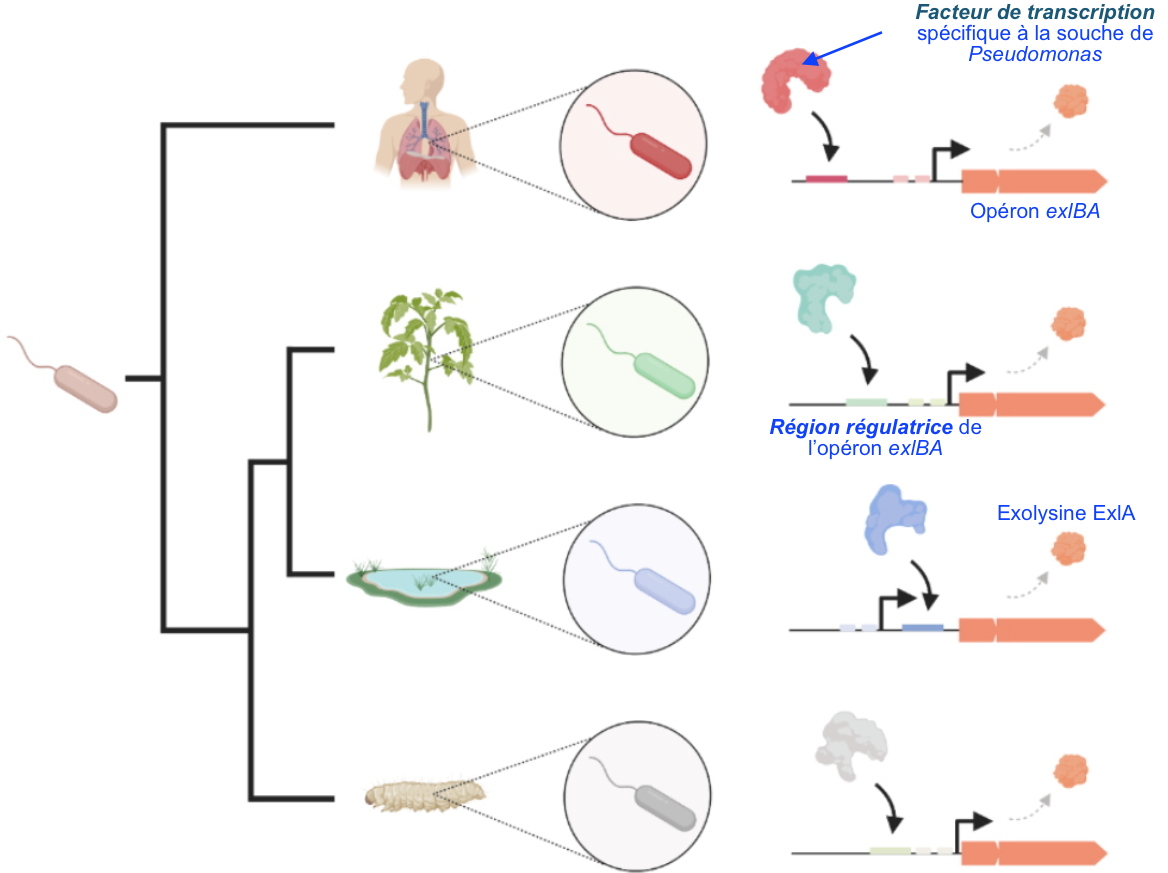
Illustration de la diversité de régulation de gènes dans des bactéries vivant dans des environnements différents.
Chaque souche a développé des séquences régulatrices spécifiques pour les gènes
exlBA permettant le recrutement de facteurs de transcription différents qui assurent une expression plus adaptée à des environnements spécifiques (poumon, plantes, eaux stagnantes, etc).
Ces travaux illustrent un mécanisme d’adaptation des bactéries à différents environnements. Après acquisition de nouveaux gènes, la pression évolutive deviendrait forte sur leurs séquences régulatrices. Ceci aurait pour conséquence de forcer la sélection de sites de fixations de régulateurs spécifiques et par conséquent une expression adaptée par les voies de signalisation préexistantes.
Un
opéron est un ensemble de gènes contigus localisés sur le chromosome bactérien et co-transcripts.
Le
transfert horizontal de gènes est un processus dans lequel un organisme intègre du matériel génétique provenant d'un autre organisme.
Un
régulon est un ensemble fonctionnel de gènes dont l'expression est co-régulée.
Un
facteur de transcription est une protéine nécessaire à l'initiation ou à la régulation de la transcription d'un gène.