La biosynthèse des ARN implique de nombreux événements biochimiques essentiels à leur maturation. D'un point de vue enzymatique, le phénomène de modification des nucléosides est certainement l'un des plus remarquable de ces événements. Il permet en effet une multitude de changements structuraux au niveau des bases canoniques adénosine, guanosine, cytosine et uridine. Un certain nombre de ces modifications, qui résultent de processus complexes (hyper-modifications), ont été caractérisées et sont principalement localisées aux positions 34 et 37 des ARN de transfert (ARNt) dans la boucle dite « anticodon » (
Figure). Elles sont particulièrement intéressantes car elles sont connues pour optimiser la fonction de décodage de l’ARN messager en contrôlant directement les interactions codon-anticodon. Les enzymes catalysant la modification des nucléosides sont des cibles fondamentales pour la recherche, car elles permettent d’étudier les interactions protéine-ARN et sont impliquées dans certaines maladies. Malgré la richesse des données génétiques, physiologiques et biochimiques de la voie de biosynthèse de la modification de l'adénine 37 de certains ARNt (voie de biosynthèse du groupement ms
2io
6A37), très peu de données structurales sont disponibles concernant les enzymes catalysant cette hyper-modification.
Depuis quelques années, des chercheurs de l’Irig étudient les enzymes impliquées dans la voie de biosynthèse conduisant à la formation du
nucléoside hyper-modifié ms
2io
6A37. Cette hyper-modification est le résultat d’une séquence de réactions enzymatiques catalysées successivement par les enzymes
MiaA,
MiaB et
MiaE, dont les deux dernières appartiennent à la classe des métallo-enzymes. En combinant des méthodes biochimique, spectroscopique et cristallographique, les chercheurs de l’Irig ont identifié les éléments structuraux de MiaE qui permettent l'amarrage de l'enzyme avec l'ARNt et la canalisation de l'oxygène moléculaire, ces deux évènements étant les primo-processus essentiels qui initient la transformation d'hydroxylation.
La structure cristallographique de l’enzyme MiaE de
Pseudomonas putida (pp-MiaE) a été obtenue par les chercheurs. Cette structure se présente sous la forme d’un domaine unique et compact qui se compose essentiellement d’un faisceau de quatre hélices-α antiparallèles abritant un centre catalytique à deux atomes de fer. Ces deux ions métalliques sont coordonnés par des histidines et des glutamates, et sont pontés par un atome d'oxygène caractérisé comme étant du type « μ-oxo » par spectroscopie. Deux canaux fonctionnels ont été identifiés. Le premier a été cartographié en utilisant des atomes de krypton (substitut de l'oxygène) introduits sous pression dans les cristaux de Pp-MiaE. Ce canal de nature hydrophobe, est incontestablement la voie d'acheminement de l'oxygène moléculaire vers le site actif pour la catalyse. Le second canal, de nature polaire, est interprété comme étant une vaste cavité permettant d’accueillir stéréo-sélectivement la partie anti-codon de l'ARNt et de positionner la base adénine 37 à proximité du site actif afin de faciliter l'hydroxylation de sa partie isopentényle. Un modèle structural du complexe MiaE-ARNt a été simulé sur la base des structures cristallographiques, des résultats biochimiques et spectroscopiques. Ce modèle révèle qu'une boucle flexible non canonique de l'enzyme MiaE, chargée positivement, est vraisemblablement impliquée dans les processus de reconnaissance et d’amarrage de l’ARNt.
L'ensemble de ces résultats ouvrent la voie à la compréhension des mécanismes impliqués dans la reconnaissance enzyme-substrat dans le cadre des réactions de modification des ARNt.
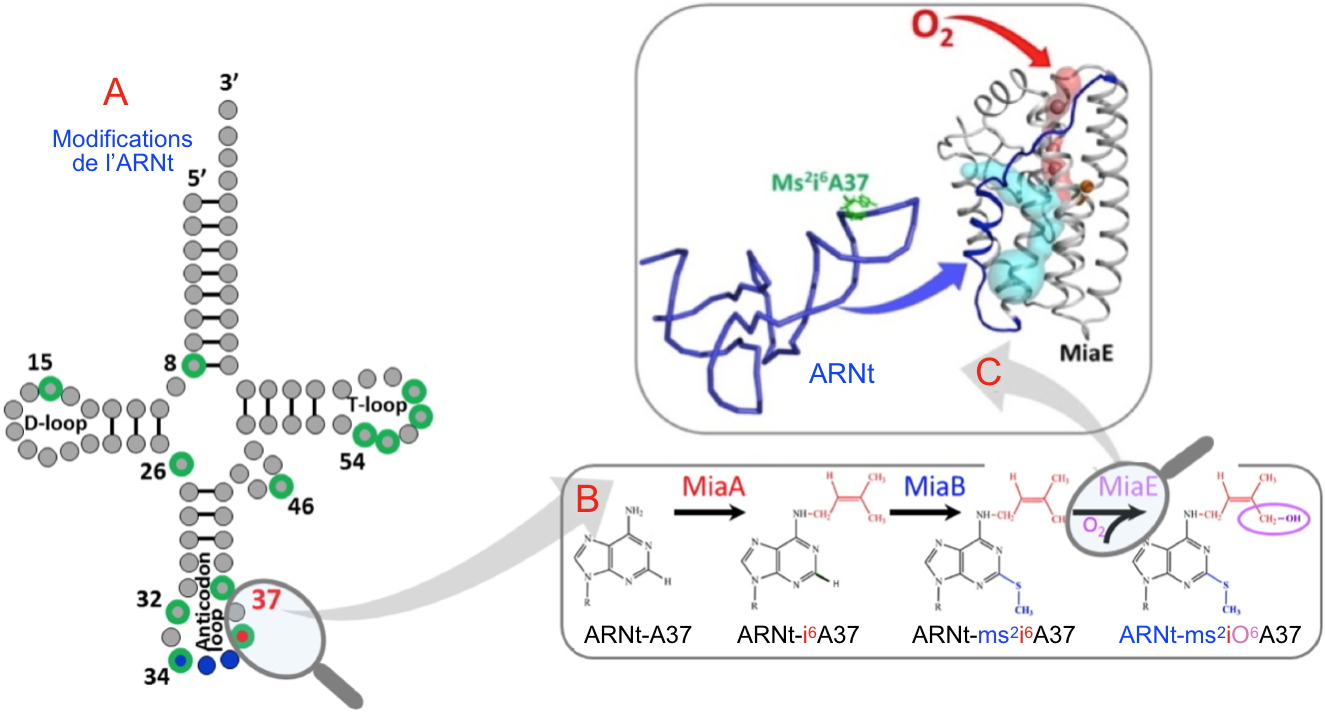 A - Modifications typiques des ARNt (en vert), dont celles en position 37 sur la boucle de l'anticodon (anticodon loop).
A - Modifications typiques des ARNt (en vert), dont celles en position 37 sur la boucle de l'anticodon (anticodon loop).
B - Détails de l’hyper-modifications A37 de l'ARNt catalysées successivement par les enzymes MiaA, MiaB et MiaE.
C - Structure de l’enzyme MiaE, le canal à oxygène est en rouge, la cavité abritant l’ARNt est en bleu claire. L'ARNt est en bleu et la base à modifiée est en vert.
Ce
nucléoside hyper-modifié est le 2-méthylthio-N6-(cis-hydroxy)isopentényl-adénosine (ms2io6A37) qui est présent en position 37 adjacente à l’anticodon de certains ARNt.
MiaA permet l'ajout du groupe isopentényl sur l'azote exocyclique N6 de l'adénosine, convertissant l'ARNt-A37 en ARNt-io6A37.
MiaB est une enzyme de la famille des Radical-SAM qui catalyse l’insertion du groupement thiométhyl en position 2, en convertissant ARNt-i6A37 en ARNt-ms2i6A37.
MiaE fait partie de la famille des protéines avec un centre à fer non-héminique. Cette famille se compose d'enzymes extrêmement diversifiées, identifiées dans tous les règnes du vivant, qui catalysent un nombre impressionnant d'oxydations chimiques différentes.