Les immunothérapies se sont récemment imposées comme un traitement de référence pour les cancers avancés, offrant globalement une amélioration de la survie des patients. Néanmoins, seule une minorité de patients en bénéficie actuellement, faute de disposer de marqueurs moléculaires fiables permettant d’identifier les répondeurs. Nous présentons dans ce travail une méthodologie
in silico exploitant les réseaux d’expression géniques afin d’identifier des signatures prédictives de la réponse à l’immunothérapie de patients atteints de cancer du rein.
Nous avons modélisé des réseaux d’expression de gènes et extrait leurs caractéristiques topologiques afin d’explorer leur pertinence pour prédire la réponse thérapeutique de patients atteints de cancer du rein. À partir d’une cohorte de plus 300 transcriptomes de tumeurs de rein, nous avons modélisé des réseaux de gène personnalisés (Figure, panel A) et étudié leur connectivité, les associations gène-gène, la similarité des réseaux et la dérégulation de voies de signalisation biologiques (Figure, panel B). Ces analyses ont permis de révéler des signatures de co-expression de gènes et d’améliorer la performance de modèles d’apprentissage automatique prédictifs de la réponse des patients à l’immunothérapie (Figure, panel C).
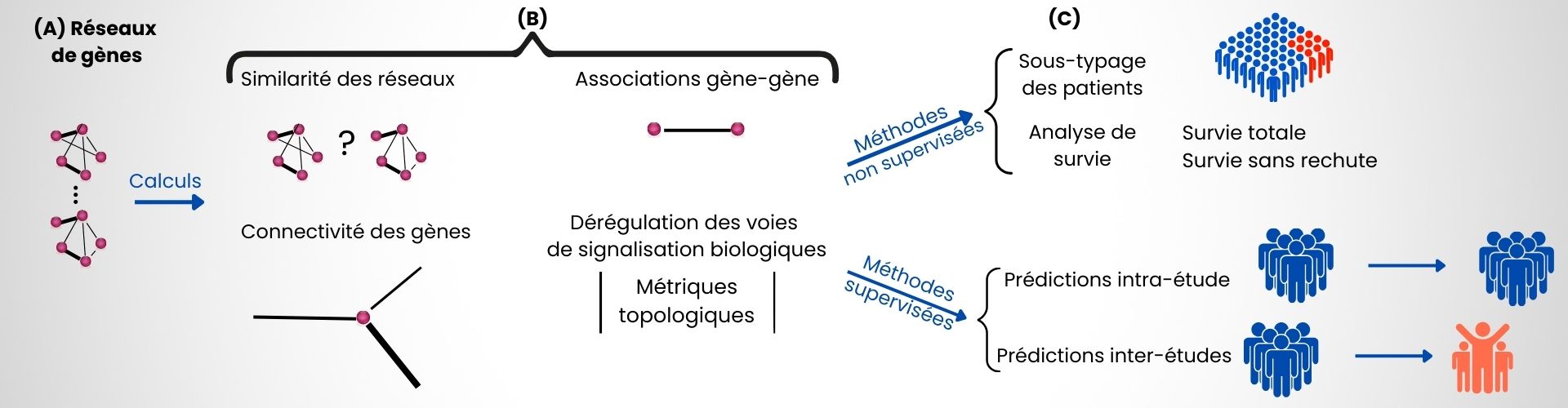
Figure : Description de la méthodologie in silico incluant (A) la modélisation de réseaux de gènes personnalisés pour chaque tumeur, (B) le calcul de quatre catégories de caractéristiques topologiques et (C) leur évaluation pour la classification des patients, l’analyse de survie et la prédiction de la réponse thérapeutique. © CEA
Nos travaux démontrent la valeur des réseaux d’expression de gènes comme source alternative de signatures moléculaires, par rapport à de simples marqueurs géniques, pour améliorer la prédiction de la réponse thérapeutique des patients atteints de cancers.
Financements
- Projets KATY et CANVAS du programme de recherche et d’innovation Horizon 2020 de l’Union européenne,
- Projet DIGPHAT de l’initiative France 2030
via le programme PEPR Santé Numérique de l’Agence Nationale pour la Recherche