La régulation de l’expression des gènes par les
microARN* (miARN) constitue un champ de recherche majeur en biologie et en médecine. Ces petites molécules non codantes contrôlent finement la production de protéines et leur dérégulation est impliquée dans de nombreuses pathologies. Comprendre précisément leur mode d’action reste toutefois un défi scientifique.
Dans une étude parue en 2025 dans Nature Communications, une équipe du CEA-Irig/Biosanté a conduit une nouvelle analyse critique de données pionnières publiées en 2019. Alors que ces premières analyses concluaient que plusieurs miARN abondants régulaient l’expression d’un grand nombre de leurs cibles, les chercheurs démontrent qu’il s’agissait d’un artefact lié à des biais méthodologiques.
En appliquant une stratégie systémique et en partageant publiquement leurs codes d’analyse, ils montrent que :
- seul un miARN, miR-92a-3p, présente une anti-corrélation robuste avec de nombreux gènes cibles prédits,
- la majorité des miARN, y compris ceux très exprimés, n’exercent pas d’effet généralisé détectable,
- les choix des paramètres d’analyse (seuils d’expression, conservation évolutive des cibles, scores de prédiction) influencent les résultats, et doivent donc être standardisés.
Au-delà de cette révision critique, le travail établit des bonnes pratiques pour l’exploitation des données de co-séquençage unicellulaire. Il insiste sur la nécessité d’évaluer la qualité des prédictions des cibles et propose d’utiliser ces données comme outils de référence pour améliorer les algorithmes de bioinformatique.
Ces avancées renforcent la fiabilité des conclusions que l’on peut tirer du co-séquençage unicellulaire, une technologie en plein essor. Elles contribueront à mieux comprendre le rôle des miARN dans les maladies complexes et à ouvrir des perspectives pour la médecine personnalisée, où les miARN apparaissent comme des biomarqueurs et des leviers thérapeutiques prometteurs.
Ce travail confère à l’équipe une compétence reconnue et rare dans l’analyse des jeux de données de co-séquençage à l’échelle de la cellule unique, renforçant son positionnement de référence dans ce domaine émergent.
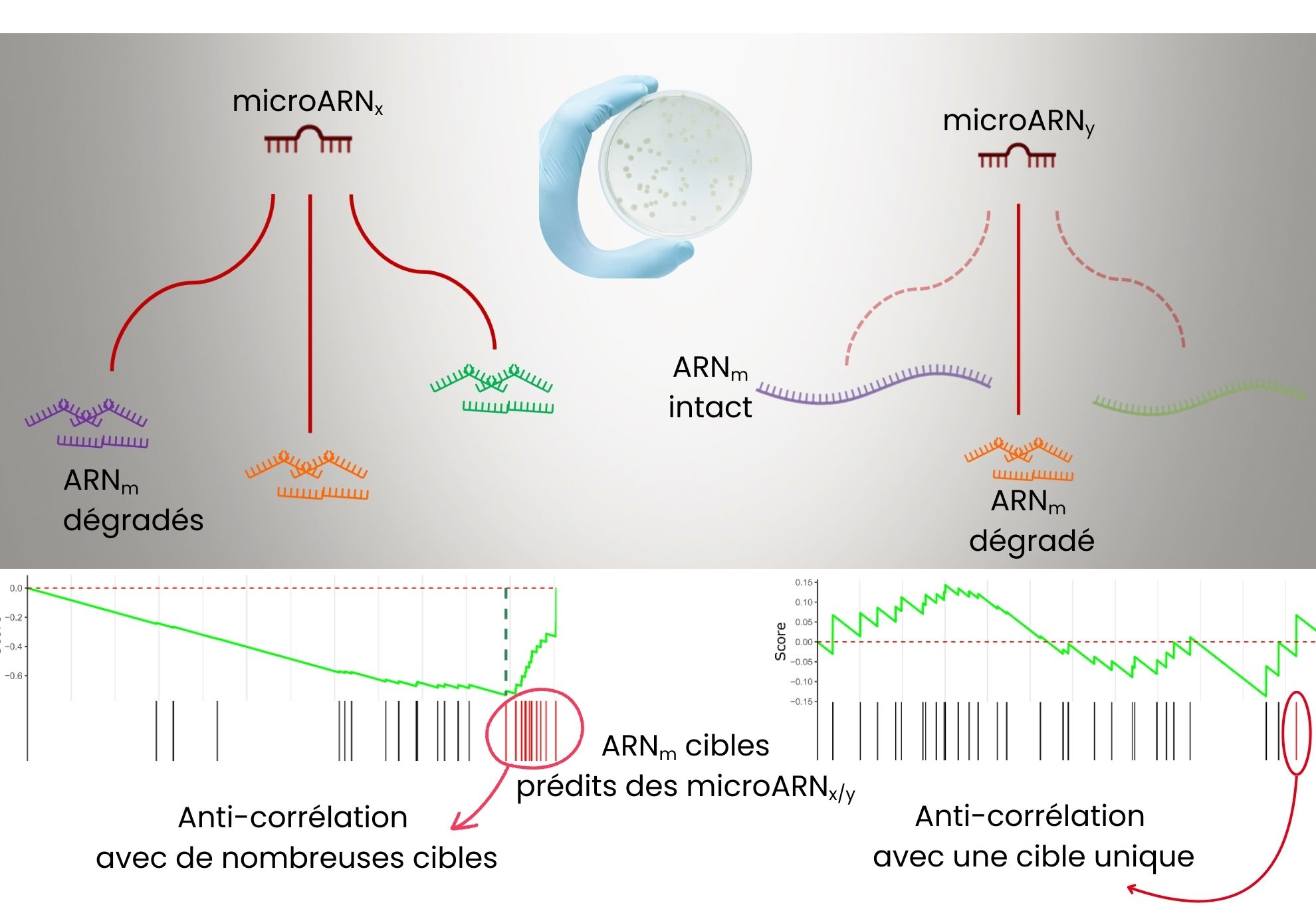
Analyse statistique de co-séquençage miARN-ARNm en cellule unique : À gauche, un microARN dégradant plusieurs cibles ARNm, identifiées à la fois par un algorithme de prédiction et par une forte anticorrélation d’expression. À droite, un microARN n’agissant que sur une seule cible, illustrant la diversité des mécanismes de régulation
Les
microARN* jouent un rôle clé dans la régulation de l’expression des gènes en dégradant un ou plusieurs ARN messagers (ARNm) cibles. Identifier précisément leurs modes d’action reste toutefois un défi, car leurs effets sont souvent subtils et contextuels.
Le co-séquençage unicellulaire, qui mesure simultanément l’expression des microARN et des ARNm dans chaque cellule, offre une approche physiologique unique pour explorer ces interactions.