Le développement de tout organisme multicellulaire (végétal ou animal) dépend de gènes dont l’activation doit se produire dans le bon tissu et au bon moment. L’activation de ces gènes est contrôlée par des protéines appelées
facteurs de transcription*. Les Auxin Response Factors (ARFs) font partie de ces facteurs: en activant ou en inhibant une multitude de gènes, ils permettent à l’auxine, une hormone végétale, de jouer des rôles toujours importants mais différents selon les tissus de la plante (par exemple orienter la croissance d’une racine en fonction de la gravité, faire émerger une fleur ou aider la tige à se diriger vers la lumière).
Comment un même signal « auxine » permet-il cette multiplicité de réponses: activation et inhibition de gènes propres à chaque tissu ?
Avec leurs collaborateurs, des chercheurs du
CEA-Irig/LPCV/Flo_Re ont tenté de comprendre comment l’auxine et les ARFs contrôlent l’activité des gènes chez Arabidopsis dans l’espace et le temps.
Chez Arabidopsis, 23 protéines ARFs sont dénombrées, certaines considérées activatrices et d’autres inhibitrices. Jusqu’à présent, il était supposé que la réponse à l’auxine dépendait des ARFs présentes dans un tissu donné, qui entraient en compétition pour activer ou réprimer chaque gène.
Mais la solution est plus complexe. En effet, les ARFs agissent en s’accrochant sur des motifs ADN présents à proximité des gènes qu’elles contrôlent. En combinant des approches de biochimie, de séquençage en cellules uniques et d’études « in planta », l’étude révèle que la diversité de réponses à l’auxine dépend aussi fortement des combinaisons de motifs présents à proximité de chaque gène.
A partir de séquences synthétiques constituées de différentes configurations de motifs, les chercheurs ont ainsi découvert que chaque ARF a une préférence pour certaines configurations, et que son activité - d’activateur ou de répresseur - varie en fonction de la disposition des motifs. L’action de l’auxine implique donc deux niveaux : d’une part, la combinaison des ARFs présentes dans chaque cellule et d’autre part, la configuration des motifs présents chez chaque gène. Cette combinaison engendre un code complexe de régulation « bi-couche » ARF/Motifs.
Ce code à deux niveaux permet à chaque gène de répondre « à sa façon » et en fonction du tissu, et joue un rôle majeur dans la diversification des réponses à l’auxine dans le développement des tissus végétaux.
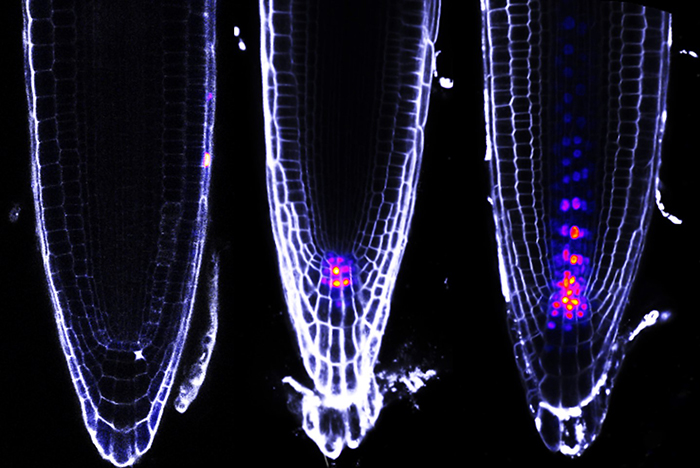
©CEA-Irig/LPCV/Flo_RE/R. Dumas
Activité dans des racines d’Arabidopsis de trois séquences d’ADN synthétiques, construites à partir de trois configurations différentes, liées par les ARFs et contrôlant l'expression d’une protéine fluorescente (mTurquoise). Cette figure illustre que des profils d’expression variés font émerger un code à double couche basé sur la composition des ARFs de chaque cellule et sur la liaison des ARFs à des configurations de motifs ADN différents. Les images ont été obtenues par microscopie confocale. La fluorescence est visualisée dans une gamme de couleurs allant du violet au jaune-orange. Images de Raquel Martin-Arevalillo.
Comprendre comment, dans un organisme, l’information d’un signal de développement est déclinée en une multitude de réponses cellulaires dans l’espace et le temps, est un élément majeur dans la compréhension du développement de cet organisme. Grâce à cette étude, il devient possible de comprendre comment l’auxine peut induire une multitude de réponses transcriptionnelles, malgré la complexité des interactions moléculaires en jeu. Cette avancée ouvre de nouvelles perspectives pour l'agriculture et la médecine, en permettant de mieux contrôler et prédire les réponses des organismes aux signaux de développement.
Facteurs de transcriptions* : protéines capables de se lier à l'ADN et permettant la régulation des gènes en activant ou en inhibant la synthèse des ARNm.
Financements : ANR ChromAuxi 18-CE12-0014-02, ERC TEMPO Project 101095380, GRAL/CBH-EUR-GS (ANR-17-EURE-0003)
Collaborations :
Laboratoire Reproduction et Développement des Plantes, Université de Lyon, ENS de Lyon, CNRS, INRAE, INRIA, 69342 Lyon, France
Institute of Synthetic Biology, University of Düsseldorf, 40225 Düsseldorf, Germany
CEPLAS – Cluster of Excellence on Plant Sciences, University of Düsseldorf, 40225 Düsseldorf, Germany
Center for Genomics and Systems Biology, New York University, New York, NY, USA
Center for Genomics and Systems Biology, New York University Abu Dhabi, Abu Dhabi, United Arab Emirates