Les données génomiques publiques connaissent une croissance exponentielle depuis ces dernières années. Leur contenu représente un fond scientifique d'envergure pour faire avancer les recherches dans différents domaines tels que la santé, l'agronomie ou encore l'écologie. Bien qu'elles contiennent jusqu'à plusieurs pétaoctets de données brutes de séquençage, ces ressources sont rarement réutilisées en raison de l'absence de moyen efficace pour interroger leurs données dans leur ensemble.
C'est dans ce contexte que des chercheurs de l'IRISA (Institut de Recherche en Informatique et Systèmes Aléatoires) de l'INRIA, en collaboration avec le Genoscope et le MIO (Institut Méditerranéen d'Océanologie), ont développé un nouvel outil informatique d'indexation et de requêtage des séquences génomiques, kmindex, dont les performances et la rapidité rendent accessibles de vastes ensembles de données génomiques.
Ils ont utilisé des k-mers (sous-séquence de longueur k extraite d'une séquence plus longue d'ADN ou d'ARN) comme élément unitaire. Lorsqu'une séquence génomique est requêtée, le nombre de k-mers partagées entre la séquence interrogée et les échantillons indexés est utilisé pour signaler des correspondances significatives. Cette solution d'indexation a ainsi permis, via la mise en place d'un serveur web ORA (Ocean Read Atlas), d'interroger plusieurs dizaines de téraoctets de données de séquence issues du projet Tara Oceans (2009-2013). À l'aide d'une ou plusieurs séquences, il a été possible d'identifier la présence de séquences similaires sous forme de carte et de graphique interactifs dans les stations de prélèvement en fonction de leurs propriétés environnementales (température, salinité, oxygène, etc.). Il a été également possible, en étudiant des variants de gènes, de voir quelles contraintes environnementales contribuent à leur évolution.
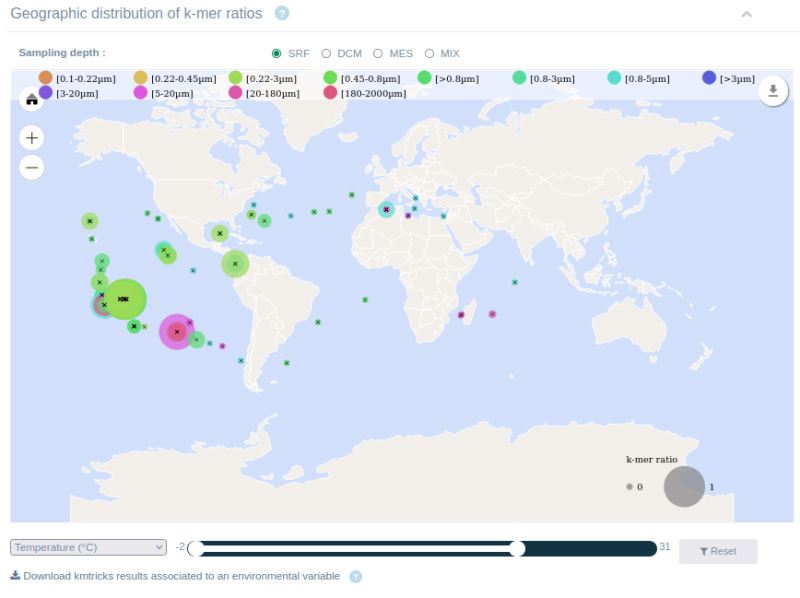
Carte de la répartition biogéographique des séquences partageant les k-mers contenus dans la séquence du gène appelé nifH de l’espèce Pseudodesulfovibrio profundus(ORA/Institut Méditerranéen d'Océanologie)
L'utilisation de kmindex dans l'interface ORA a réduit de plusieurs ordres de grandeur le temps de traitement des requêtes tout en maintenant la qualité des résultats et en réduisant l'obtention de faux positifs.
Grâce à cette étude, publiée dans le journal Nature Computational Science, un nouveau jalon a été franchi concernant la prise en charge de projets génomiques d'envergure. Les fonctionnalités de ce nouvel outil d'indexation, kmindex, permettent de rendre accessible à la communauté scientifique de très grande quantités de données de séquençage.
Voir aussi
Actualité CNRS Terre et Univers
Expédition Tara Océan : un Google de la génomique pour traiter la quantité de données collectées (Article Le Monde réservé aux abonnés)