Si les récents développements en « omique » à résolution spatiale sont en train de révolutionner l'exploration de la complexité tissulaire, la plupart des solutions disponibles se concentrent néanmoins uniquement sur la détection de la présence d'ARN. Des études ayant montré que la demi-vie de ces molécules peut varier de 40 minutes à 9 heures, un test de profilage transcriptomique ne reflète pas directement l'état transcriptionnel de la cellule au moment de la mesure. De plus, par définition, une analyse transcriptomique ne peut identifier que les lectures des gènes exprimés, mais ne peut pas distinguer les gènes réprimés des gènes maintenus dans un état transcriptionnel favorable, car les deux sont transcriptionnellement inactifs.
L'utilisation de stratégies d'immunoprécipitation de la chromatine (utilisation d'anticorps pour capturer des protéines associées à l'ADN) pour repérer des modifications d'histones (protéines qui compactent l'ADN) dans le génome est devenue la méthode de référence pour évaluer l'état de régulation de la transcription à l'instant « t » dans des échantillons. Cette stratégie, plus récemment déclinée dans sa version enzymatique « Cut&Tag » a permis un gain de sensibilité en réduisant le nombre de cellules requises, de quelques millions à quelques milliers, atteignant même l'échelle de la cellule unique. Ainsi, il est possible d'identifier des histones ayant des modifications associées à l'état de régulation de l'expression de gènes.
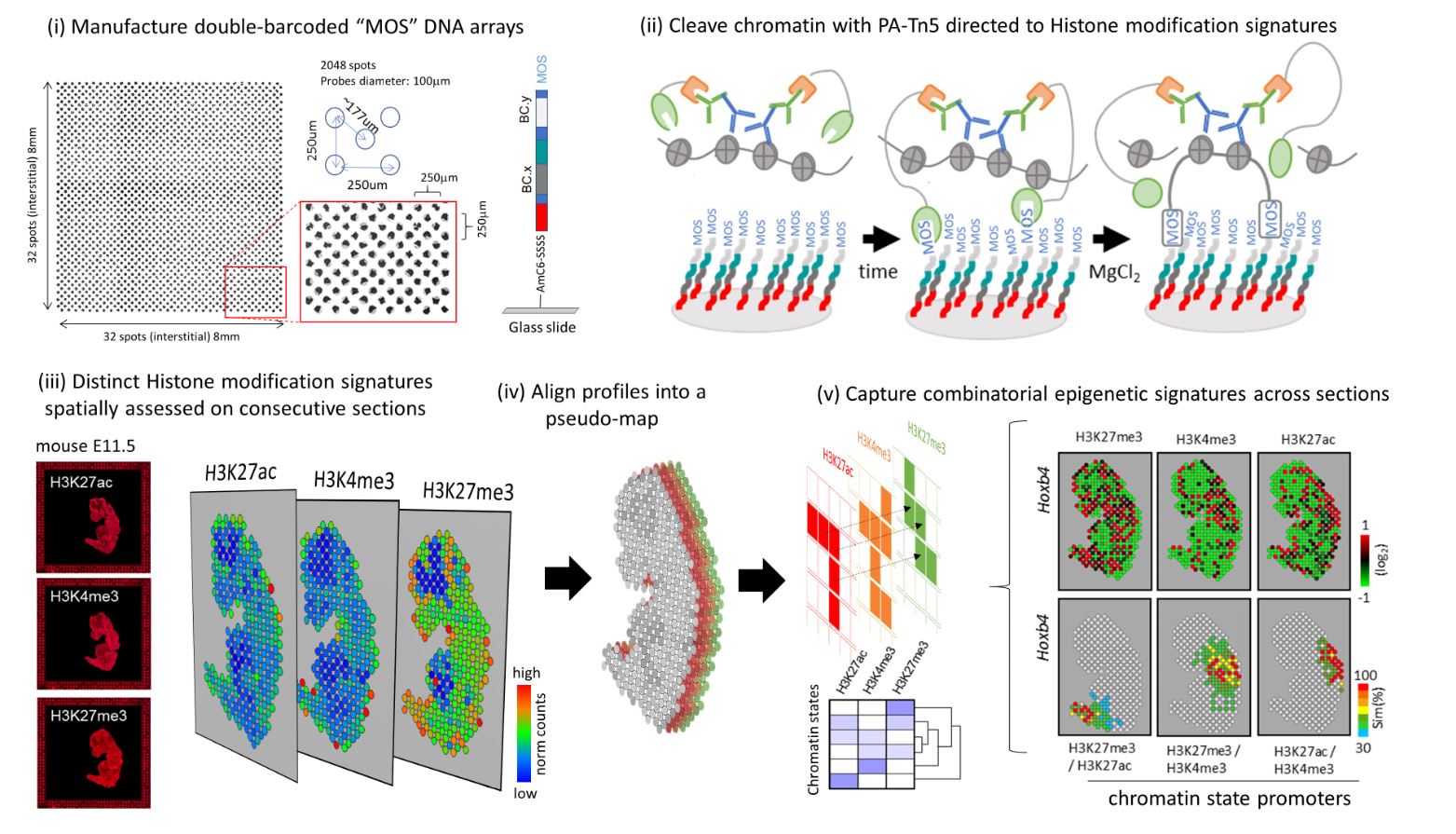
Des chercheurs du Genoscope ont mis au point une nouvelle méthode permettant d'identifier des signatures de modification d'histones dans l'espace grâce à une puce d'ADN présentant des codes-barres moléculaires uniques à une résolution de 100 micromètres. Cette technologie, basée sur l'utilisation de la protéine recombinante PA-Tn5, permet de cliver la chromatine et de l'immobiliser par la création d'un pont covalent avec les sondes d'ADN de la puce. Ainsi, les chercheurs ont validé cette méthodologie sur des cerveaux de souris adultes, sur des embryons de souris congelés, ainsi que sur du matériel biologique osseux préalablement décalcifié et conservé dans des blocs de paraffine (FFPE), condition dans laquelle l'ARN est mal conservé, d'autant plus après des procédures de décalcification.
Une première technologie a été développée par une équipe de l'Université de Yale aux Etats-Unis, pour cartographier les modifications d'histones sur des section de tissues, utilisant des canaux microfluidiques de 20 microns (Deng et al ; Science 2022). La technique mise au point par les chercheurs du Genoscope n'est pas aussi résolutive que la première, mais la flexibilité de fabrication de puces d'ADN couvrant des surfaces personnalisées à moindre coût, grâce au nombre combinatoire de sondes requises, la rend extrêmement performante pour l'analyse d'un grand nombre de coupes de tissus, même de grandes dimensions.
@ M. Mendoza-Parra / CEA
Dans cette étude publiée dans Genome Research, l'équipe du Genoscope a également souligné la nécessité de collecter des coupes consécutives pour acquérir des signatures distinctes de modifications d'histones, lesquelles peuvent ensuite être combinées afin de révéler les signatures d'état de la chromatine et de les comparer dans un contexte spatial. Cette stratégie devrait gagner en puissance dans les années à venir grâce à une diminution des coûts de séquençage, permettant ainsi de collecter plusieurs coupes consécutives pour reconstruire des vues volumétriques des signatures d'état de la chromatine qui régissent l'architecture tissulaire.
Contact : mmendoza@genoscope.cns.fr