La paramécie, organisme modèle des eucaryotes unicellulaires
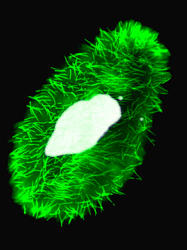
Les cils de la paramécie, ici marqués en vert, recouvrent toute la cellule et permettent à l’organisme de nager à la recherche des bactéries dont il se nourrit. A droite de l’énorme masse du macronoyau, marqué en bleu clair, on distingue les micronoyaux sous la forme de deux petits points. (Photo J. Beisson)
Bien connue des collégiens comme modèle d’organisme unicellulaire, la paramécie (Paramecium tetraurelia) est une cellule eucaryote de grande taille (120 micromètres), recouverte de cils vibratiles, qui appartient au phylum des ciliés (Ciliophora). Les ciliés sont apparentés, au sein du clade des alvéolés, avec le groupe des apicomplexa, unicellulaires parasites parmi lesquels on trouvePlasmodium falciparum, principal agent du paludisme. La paramécie est un organisme à la fois unicellulaire et complexe ; elle constitue par conséquent un excellent modèle pour l’étude, par une approche génétique, de nombreuses fonctions différenciées présentes chez les organismes multicellulaires mais absentes chez des eucaryotes unicellulaires plus simples comme la levure.
Les ciliés présentent la particularité fascinante de séparer les lignées germinale et somatique à l’intérieur d’un cytoplasme unique. Ces cellules possèdent en effet deux noyaux. Un noyau germinal (le micronoyau) assure la transmission de l’information génétique à travers les processus sexuels, tandis qu’un noyau somatique (le macronoyau) assure l’expression de cette information. A chaque génération sexuelle, un nouveau noyau somatique est produit par des réarrangements programmés de tout le génome contenu dans le noyau germinal.
Des processus fondamentaux sont étudiés chez la paramécie
-
Ces réarrangements programmés du génome, ainsi que l’amplification massive d’ADN qui produit le noyau somatique, impliquent des événements de fragmentation chromosomique, d’addition de télomères et d’élimination précise de séquences internes. Ces processus sont similaires aux réarrangements somatiques intervenant dans la genèse de la diversité des immunoglobulines, et aux remaniements chromosomiques associés à certains cancers chez les vertébrés. Chez la paramécie, ces réarrangements chromosomiques touchent l’ensemble du génome et peuvent être facilement déclenchés.
-
L’extinction génique dépendante de l’homologie de séquence, mécanisme de défense des génomes contre des acides nucléiques parasites, est présent chez la paramécie et a été conservé au cours de l’évolution des eucaryotes. Ce mécanisme, appelé aussi interférence ARN, peut être expérimentalement déclenché par l’introduction d’un transgène ou d’ARN double brin dans la cellule. Il agit au niveau post-transcriptionnel et conduit à la dégradation de tous les ARN messagers homologues dans la cellule. L’interférence ARN est aussi impliquée dans le développement du noyau somatique de la paramécie, ce qui peut expliquer l’hérédité non mendélienne de certains caractères chez ce cilié. La paramécie est un organisme privilégié pour l’étude de ces phénomènes épigénétiques, car ils y sont facilement accessibles à l’expérimentation. Leur rôle biologique, par exemple dans le développement des eucaryotes supérieurs, reste à analyser.
-
La paramécie est également capable d’une exocytose régulée de vésicules de sécrétion (trichocystes) en réponse à une stimulation externe, comme cela se produit lors de la sécrétion d’hormones ou de neuromédiateurs chez les animaux. Cette sécrétion régulée implique une étape de fusion membranaire déclenchée par une voie de signalisation trans-membranaire. Elle sert à la défense contre les prédateurs chez la paramécie, mais n’est pas essentielle dans les conditions de culture du laboratoire. En conséquence, la paramécie fournit l’un des rares modèles où la sécrétion régulée, et en particulier la dernière étape de fusion membranaire lors de l’exocytose, peut-être disséquée par une approche génétique.
-
La membrane de la paramécie est excitable. Comme dans les neurones et les cellules musculaires, cette excitabilité membranaire est gouvernée par l’activité de canaux ioniques et de récepteurs membranaires. Chez la paramécie, leur fonctionnement contrôle la vitesse et la direction de battement des cils de la cellule. L’activité de ces canaux est donc facilement visualisée par un comportement de nage. C’est chez la paramécie qu’ont été isolés les premiers mutants de canaux ioniques à la fin des années 1960, ainsi que les premiers mutants de calmoduline (protéine ubiquitaire réglant de nombreux processus cellulaires en réponse à des changements de concentration en calcium).
-
La paramécie possède des corps basaux, organites homologues des centrioles présents dans les cellules des organismes multicellulaires. Ces édifices protéiques complexes ont la remarquable propriété de se dupliquer à chaque division par un mécanisme non encore élucidé. La duplication des corps basaux joue un rôle clé dans la division et l’organisation cellulaires, comme dans la morphogenèse de la paramécie. La dissection génétique de ce phénomène, pour lequel la paramécie constitue un modèle extrêmement avantageux, a permis la découverte de nouvelles protéines.
Deux outils essentiels disponibles chez la paramécie : une génétique performante...

L’utilisation d’anticorps anti-tubuline marqués fait ici apparaître les corps basaux de la paramécie, situés à la base des cils. Le macronoyau et les micronoyaux (deux petits points collés à droite du macronoyau) apparaissent de nouveau en bleu. (Photo F. Ruiz)
Plus de 50 ans de génétique classique ont permis d’accumuler près de 200 mutants mendéliens de paramécie affectés dans des processus cellulaires très divers (morphogenèse, sécrétion régulée, cycle cellulaire, variation antigénique, détermination et expression du type sexuel, réarrangements du génome). En effet, la paramécie se prête admirablement à l’analyse génétique en raison de ses deux modes de reproduction sexuée, l’autogamie et la conjugaison. L’autogamie est un processus d’autofécondation qui rend le génome zygotique entièrement homozygote en une génération. Les souches stockées sont ainsi manipulables comme des haplo&iauml;des. La conjugaison est un processus de fécondation réciproque aboutissant à la formation de noyaux zygotiques identiques dans les deux partenaires, ce qui permet d’identifier très facilement les caractères à hérédité mendélienne et de les distinguer des caractères à hérédité maternelle. Les gènes identifiés par mutation peuvent être clonés par complémentation fonctionnelle.
... et une génomique fonctionnelle par interférence ARN
L’extinction génique, provoquée par l’introduction de transgènes, fournit un outil puissant pour l’analyse fonctionnelle des gènes. Toutefois, l’outil idéal est l’interférence ARN, obtenue avec une remarquable efficacité chez la paramécie par ingestion de bactéries produisant de l’ARN double brin. Cette méthode de « feeding », originalement développée chez le nématode Caenorhabditis elegans, permet d’envisager une analyse fonctionnelle à grande échelle des ORFs identifiées.
La paramécie et « ses » génomes
Les noyaux de la paramécie, micronoyau et macronoyau, sont différents tant par leur structure que par leur fonction. Le micronoyau (présent en deux exemplaires chez l’espèce P. tetraurelia) est diploïde, représente la lignée germinale et est totalement silencieux pour la transcription. C’est lui qui subit la méiose et la fécondation lors des événements sexuels (conjugaison entre cellules compétentes, autogamie au sein d’une même cellule).
Le macronoyau, hautement polyploïde (environ 1000 n), représente la lignée somatique et est le siège de la transcription. Le macronoyau comme le micronoyau dérivent de copies du noyau zygotique. Le développement programmé du macronoyau comprend une amplification de l’ADN par un facteur d’environ 250, l’élimination précise de courtes séquences internes appelées IES et l’élimination imprécise de régions riches en transposons et en séquences répétées, vraisemblablement hétérochromatiques, ce qui aboutit à la fragmentation des chromosomes. Les extrémités ainsi créées sont réparées par ajout de télomères.
Propriétés comparées du micronoyau et du macronoyau
| Micronoyau | Macronoyau |
| Ploïdie | 2 n | 1000 n
|
| Taille du génome | 100 à 120 mégabases | 80 à 90% de la complexité micronucléaire |
| Nombre de chromosomes | 50 | 350 |
| Taille des chromosomes | 2 mégabases | 300 (50 à 1000) kilobases |
La grande majorité de l’hétérochromatine est éliminée lors du développement du macronoyau ; celui-ci est donc essentiellement euchromatique et dépourvu de séquences répétées et de microsatellites. Ceci représente un avantage certain pour un premier projet de séquençage du génome, parce que la présence de séquences répétées constitue une difficulté technique, notamment lors de l’étape d’assemblage du génome.
Grâce notamment à cette élimination des séquences répétées, le génome macronucléaire de la paramécie est très « compact » : on estime que sa fraction codante est supérieure à 70% (dans le génome humain, elle est de l’ordre de 1% !). Les introns sont uniformément petits (de 18 à 35 bases) et les régions intergéniques, généralement inférieures à 50 100 bases, peuvent n’être longues que de quelques bases (minimum de 9 bases observé jusqu’à présent). Cette remarquable compacité fait du macronoyau un matériel de choix pour l’inventaire des gènes de la paramécie.
Cet inventaire bénéficiera sans doute aussi du projet de séquençage du génome deTetrahymena thermophila, un autre cilié évolutivement distant de plus de 100 millions d’années de la paramécie, et sur lequel travaille également une vaste communauté de chercheurs.
Historique du projet de séquençage de la paramécie
-
Août 1999 : à l’initiative du laboratoire "Génétique de la Dynamique Cellulaire chez la Paramécie" au Centre de Génétique Moléculaire du CNRS de Gif-sur-Yvette, un Consortium de 11 laboratoires européens et nord américains s’est créé pour autofinancer un projet pilote de séquençage aléatoire du génome de la paramécie. Ce financement a permis le séquençage de 3000 extrémités d’inserts d’une banque d’ADN du macronoyau. Les séquences annotées ont été déposées en novembre 2000 dans la division GSS des bases de données publiques. (P. Dessen
et al. 2001, Trends Genet 17 : 306-308 ; L. Sperling
et
al. 2002, Euk Cell 1:341-352).
-
Octobre 2000 : réunion des membres européens de ce Consortium à Varsovie pour définir la suite à donner au projet pilote. Il a été notamment décidé d’isoler et de séquencer un chromosome du macronoyau long d’une mégabase. Ce travail a été commencé fin 2001 et est en voie de finition à la mi-2003.
-
Juillet 2002 : le CNRS soutient la communauté scientifique européenne en créant un Groupement De Recherche Européen (GDRE), structure dotée d’un financement permettant les réunions et les échanges en son sein. Une première réunion s’est tenue à Dourdan en Octobre 2002.
-
Octobre 2002 : le CNRS soutient le projet génome de la paramécie. Il alloue un budget de 100 000 Euros, dans le cadre de son programme de séquençage à grande échelle, pour amorcer un séquençage de l’ADN du macronoyau de la paramécie au Genoscope.
-
Avril 2003 : le Conseil Scientifique du Genoscope approuve formellement la réalisation par le Genoscope, sur ses fonds propres, du séquençage total de l’ADN du macronoyau de la paramécie.