Les photolyases de l'ADN sont des flavoenzymes photoactives largement répandues dans tous les règnes du vivant. Elles réparent les principales lésions induites dans l'ADN par les UV : les dimères cyclobutaniques de pyrimidines (CPDs) et les photoproduits 6-4 (6-4 PPs). Selon leur structure, les photolyases spécifiques de la lésion CPD sont réparties en trois classes, I-III. Seules les photolyases de classe I (bactériennes) ont été étudiées de manière approfondie. Les photolyases de classe II (retrouvées chez les procaryotes et les eucaryotes, y compris les plantes et la plupart des animaux supérieurs) et de classe III ont été découvertes plus récemment et le mécanisme exact de leur fonctionnement reste encore à élucider.
Un élément fonctionnel important pour toutes les photolyases est une chaine de transfert d'électrons composée typiquement de trois résidus tryptophane qui permet de réduire leur cofacteur flavine adénine dinucléotide (FAD) excité par la lumière bleue (ou UVA). Cette réaction nommée "photoactivation" est préalable à la photoréparation de l'ADN et est essentielle car seule une photolyase avec la flavine complètement réduite en FADH− peut catalyser la réparation de l'ADN endommagé.
Dans cette étude de la photolyase classe II de l'archéobactérie Methanosarcina mazei, les auteurs ont montré que le FAD photoexcité est réduit via la chaîne de tryptophanes W381−W360−W388 (Figure 1), en produisant une paire de radicaux FADŸ- W388HŸ+. W388HŸ+ est stabilisé par déprotonation en 350 picosecondes, soit mille fois plus rapidement que la déprotonation des cations radicalaires WHŸ+ dans les autres photolyases ou encore celle des WHŸ+ libres en solution aqueuse. (Figure 1)
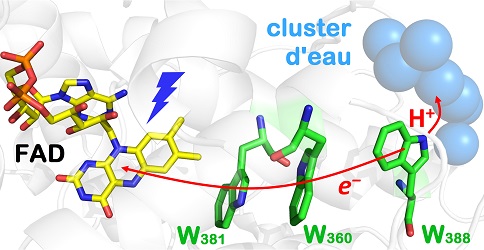
Figure 1 : La chaine de transfert d'électron dans la photolyase classe II de l'archéobactérie Methanosarcina mazei.
Des mutations d'accepteurs intramoléculaires possibles du proton (acides aminés dans la proximité de W388) se sont avérées sans effet. En examinant la structure cristallographique de l'enzyme, les chercheurs ont identifié un cluster de molécules d'eau spécifique et unique des photolyases classe II. Ce cluster est lié à la protéine et l'une de ses molécules d'eau est idéalement positionnée pour accepter le proton de W388HŸ+. La vitesse exceptionnelle de déprotonation empêche la recombinaison des radicaux et assure une photoactivation efficace.
Les résultats de cette nouvelle étude permettent d'ajouter les photolyases de classe II aux systèmes biologiques dans lesquels des réseaux de molécules d'eau jouent un rôle crucial dans le transfert des protons, comme par exemple les bactériorhodopsines. Des simulations de dynamique moléculaire suite à la formation du Trp388HŸ+ devraient permettre de mieux saisir l'efficacité et la rapidité inattendues du transfert de proton vers le cluster d'eau. Une meilleure compréhension du transfert de charge dans la photolyase pourrait inspirer la conception de systèmes artificiels nécessitant une séparation de charges rapide et efficace, tels que ceux que l'on développe pour la photosynthèse artificielle.