Responsable
Muriel GONDRY
muriel.gondry@cea.fr / muriel.gondry@i2bc.paris-saclay.fr
Tél : +33 1 69 08 76 47![Appeler : +33 1 69 08 76 47]()

De gauche à droite : P. Belin, I.B. Jacques, E. Favry, M. Gondry, J. Seguin, M. Moutiez
 Cette équipe fait partie de l'I2BC (Enzymologie et Biosynthèse Peptidique Non Ribosomale)
Cette équipe fait partie de l'I2BC (Enzymologie et Biosynthèse Peptidique Non Ribosomale)
Les recherches en cours concernent la synthèse non
ribosomale de produits naturels peptidiques, et, plus particulièrement,
des cyclodipeptides et de leurs dérivés complexes, les dicétopipérazines
(DKP). Les DKP constituent une large classe de produits naturels
synthétisés essentiellement par des microorganismes. Leurs rôles
physiologiques sont encore peu connus mais certaines DKP seraient
impliquées dans des processus de communication cellulaire ; quant à
leurs activités pharmacologiques, elles sont souvent remarquables et
diverses telles qu'antibactériennes, antivirales ou anticancéreuses
(Figure 1).
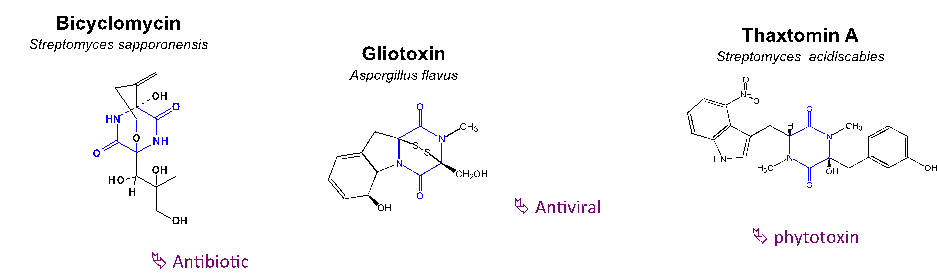 Figure 1 : Exemples de DKP bioactifs. Le noyau cyclodipeptidique est en bleu.
Figure 1 : Exemples de DKP bioactifs. Le noyau cyclodipeptidique est en bleu.
Identification et décryptage de nouvelles voies de biosynthèse de petites molécules peptidiques
Bon nombre de peptides bioactifs sont synthétisés indépendamment du ribosome par des enzymes dédiées. La majorité de ces peptides non ribosomaux sont produits par de larges enzymes multimodulaires, les NRPS (Non Ribosomal Peptide Synthetase). Cependant, l'équipe, en collaboration avec l'équipe de J.-L. Pernodet (Equipe MMA, Département de Microbiologie, I2BC), a identifié une nouvelle famille d'enzymes, les synthases de cyclodipeptides (CDPS) qui produisent des cyclodipeptides variés. Ces enzymes sont souvent associées à des enzymes de modification des cyclodipeptides dans des voies de biosynthèse dévolues à la production de DKP (Figure 2). Un des objectifs de notre équipe est de poursuivre cette identification de nouvelles voies de biosynthèse et de décrypter la diversité des molécules synthétisées par ces voies de biosynthèse.

Figure 2 : Quelques exemples de voies de
biosynthèse de DKP associant des CDPS (gènes en marron) et des enzymes de
modification des cyclodipeptides (gènes en rouge : P450 ; en
bleu : méthyltransférases ; en violet : diverses oxygénases)
chez différents organismes.
Caractérisation fonctionnelle et structurale des enzymes de biosynthèse
La caractérisation approfondie des CDPS est en cours au laboratoire. Les études ont déjà permis de montrer que ces enzymes détournent des ARNt aminoacylés (aa-ARNt) de leur rôle canonique dans la synthèse protéique ribosomale pour les utiliser comme substrats et catalyser la formation de cyclodipeptides variés. Nous avons également mis en évidence une similitude structurale importante des CDPS avec les aminoacyl-ARNt synthétases de classe I, enzymes qui catalysent le chargement des acides aminés sur leurs ARNt correspondants pour former les aa-ARNt (Figure 3).
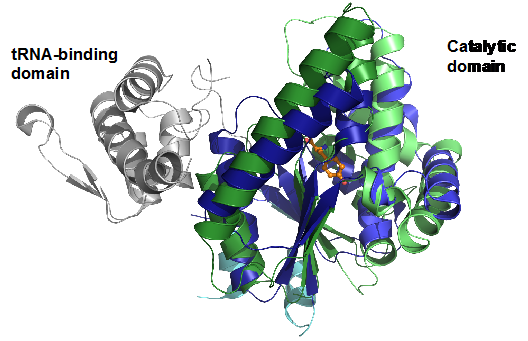
Figure 3 : Superposition de la structure de la
CDPS AlbC (en vert) et de la TyrRS, avec son domaine catalytique (bleu), son domaine
de liaison de l’ARNt (gris) et la Tyr substrat (orange).
Récemment, nous avons élucidé le mécanisme catalytique emprunté par les CDPS qui est de type ping-pong et transite par la formation successive de deux intermédiaires covalents, un aminoacyl-enzyme puis un dipeptidyl-enzyme (Figure 4). Nous avons aussi identifié de premiers déterminants de la spécificité des CDPS et ce travail se poursuit actuellement au laboratoire.
Nous nous intéressons également aux enzymes de modification des cyclodipeptides qui sont associées aux CDPS dans les voies de biosynthèse de DKP. Ainsi, nous avons étudié CYP121, un cytochrome P450 de Mycobacterium tuberculosis, qui a été décrit comme essentiel à la viabilité du pathogène. Notre travail a conduit à l'identification du cyclodipeptide substrat et du produit de la réaction catalysée par CYP121 ainsi qu'à la détermination de sa spécificité de substrat et de réaction. Par ailleurs, comme CYP121 est une nouvelle cible thérapeutique potentielle, nous développons des analogues de substrat inhibiteurs sélectifs de CYP121.
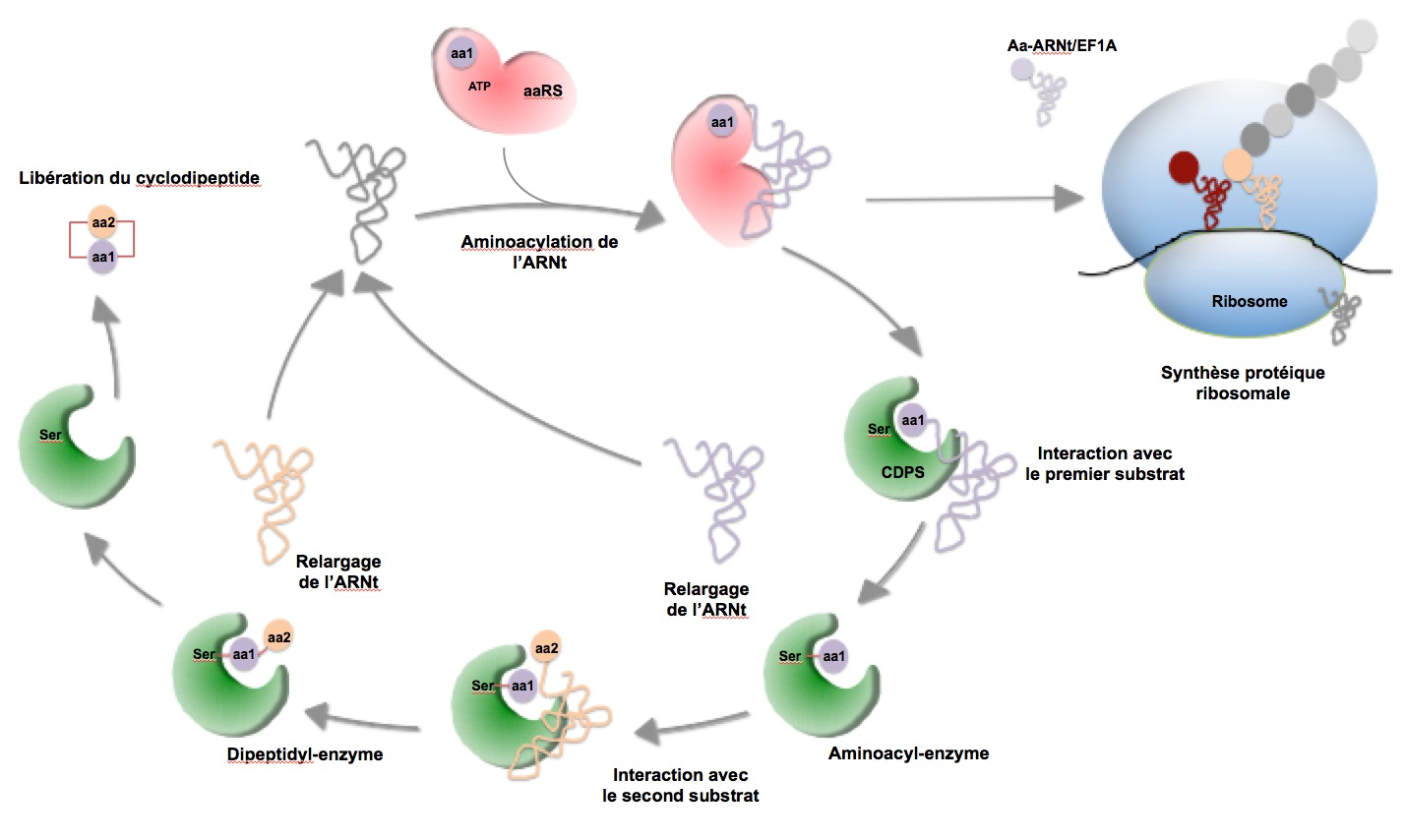 Figure 4 : Le mécanisme global emprunté par les CDPS. Les ARNt sont aminoacylés par les aaRS (en rouge) et sont délivrés au ribosome (en bleu) pour la synthèse protéique ribosomale. Des aa-ARNt sont détournés par les CDPS (en vert) qui les utilisent comme substrats via un mécanisme ping-pong.
Figure 4 : Le mécanisme global emprunté par les CDPS. Les ARNt sont aminoacylés par les aaRS (en rouge) et sont délivrés au ribosome (en bleu) pour la synthèse protéique ribosomale. Des aa-ARNt sont détournés par les CDPS (en vert) qui les utilisent comme substrats via un mécanisme ping-pong.
Ingénierie des voies de biosynthèse des peptides non ribosomaux
Les approches d'ingénierie nécessitent de parfaire nos connaissances sur la spécificité des CDPS et des enzymes de modification, en particulier pour l'obtention de nouvelles voies de biosynthèse capables d'incorporer des acides aminés non protéinogéniques. Ce travail est actuellement en cours au laboratoire tant au niveau des CDPS que des enzymes de modification.