Pour répondre aux besoins de diagnostic clinique et de dépistage microbiologique en général, il est essentiel de pouvoir identifier avec rapidité et fiabilité les microorganismes présents dans un échantillon de microbiote (flore microbienne normale ou pathologique). Afin de mieux appréhender les constituants individuels des microbiotes, la culturomique se développe comme moyen de générer de nombreux isolats microbiens présentant des caractéristiques très distinctes en testant systématiquement des centaines de conditions de culture différentes. Des méthodes d'identification à haut débit doivent maintenant être développées pour cribler rapidement ces isolats.
Dans la présente validation de concept, les chercheurs proposent une méthodologie innovante de protéotypage multiplex pour l'analyse simultanée de 21 échantillons bactériens par spectrométrie de masse en tandem, sans s'appuyer sur un marquage différentiel des peptides qui aurait recours à des réactifs chimiques couteux. La méthode repose sur le pré-fractionnement des peptidomes de chaque isolat microbien par chromatographie en phase inverse, le mélange de fractions spécifiques de chaque isolat, différencié selon son hydrophobicité, suivis de l'analyse du mélange en une seule expérience de nanoLC-MS/MS (“Liquid Chromatography coupled to tandem Mass Spectrometry"). Les signaux propres à chaque organisme sont distingués des autres signaux par leurs propriétés hydrophobes et le procédé a permis d'identifier les 21 microorganismes en une seule analyse de 60 minutes. Les chercheurs ont également développé un concept de protéotypage spécifique afin d'attribuer des informations taxonomiques à des signaux restreints par unité de temps grâce à des traitements informatiques astucieux de phylopeptidomique.
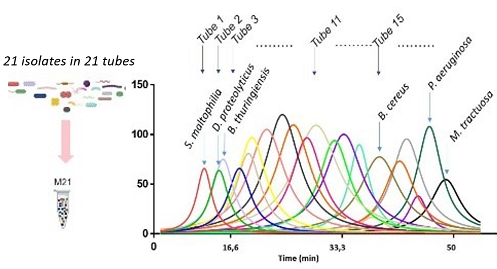
Schéma du protocole expérimental et résultat d'une analyse © J Armengaud / CEA
En développant une stratégie de multiplexage sans étiquette pour l'analyse d'isolats microbiens par spectrométrie de masse en tandem et au vu des progrès technologiques récents de cette approche, les auteurs de cette étude envisagent de pouvoir identifier jusqu'à 200 isolats microbiens par heure, de quoi révolutionner la "culturomique" et faire progresser notre connaissance des microbiotes.
Contacts : Béatrice Alpha-Bazin (beatrice.alpha-bazin@cea.fr) – Jean Armengaud (jean.armengaud@cea.fr)
Voir également : Institut des sciences du vivant Frédéric Joliot - Identification rapide et à grande échelle du microbiote pulmonaire de la mucoviscidose par métaprotéomique (cea.fr)
- Le protéotypage repose sur l'analyse et l'identification des peptides par spectrométrie de masse.
- Le microbiote est l'ensemble des micro-organismes – bactéries, virus, parasites et champignons non pathogènes, dits commensaux – qui vivent dans un environnement spécifique. Chez l'Homme le microbiote intestinal étant le plus « peuplé » d'entre eux, abritant 10
12 à 10
14 micro-organismes
- La phylopeptidomique est une méthode mathématique développée au Li2D pour identifier la contribution de chacun des organismes qui composent un microbiote dans un jeu de données de métaprotéomique. Voir
Phylopeptidomique : un nouvel outil d'analyse des microbiotes - La culturomique consiste en la multiplication des milieux et conditions de culture et en l'identification rapide de bactéries par spectrométrie de masse. Elle a notamment permis la découverte d'espèces jamais isolées chez l'homme ainsi que de nouvelles espèces ou genres bactériens. Elle est en cela très complémentaire de la métagénomique qui évalue relativement bien la présence des différentes flores majoritaires, notamment de bactéries non encore cultivables, quand la culturomique évalue mieux la diversité des flores minoritaires, dominées par les quelques espèces surreprésentées.