L'enzyme NadA intéresse les scientifiques car elle intervient dans la synthèse d'un cofacteur essentiel dans le métabolisme d'organismes pathogènes, comme Helicobacter pyloriou Microbacterium leprae. Le premier est une bactérie liée à l'ulcère et au cancer de l'estomac, tandis que le second est responsable de la lèpre. Grâce à une technique de cristallographie en atmosphère contrôlée, des chercheurs du CEA-IBS et du CEA-BIG ont décrypté le mécanisme catalytique de cette enzyme dans des conditions anaérobies (sans oxygène) nécessaires à son fonctionnement et proches de celles présentes dans un estomac.
« Nous avons observé un agrégat, composé de quatre atomes de fer et de quatre atomes de soufre, qui est le siège d'une réaction catalytique importante pour la fonction de l'enzyme », explique Juan C. Fontecilla-Camps, chercheur au CEA-IBS. Pour cela, deux mutants du site catalytique ont été fabriqués : l'un pour arrêter la réaction de catalyse au niveau d'un intermédiaire (qui normalement est instable) et l'autre pour piéger le produit de la réaction. « Nous avons pu suivre la réaction d'un point de vue structural grâce à l'analyse de l'intermédiaire et du produit », poursuit le scientifique. Avec quelles observations ? « Le produit se réoriente dans le centre catalytique de la protéine. C'est un résultat inattendu pour cette enzyme. » Les chercheurs souhaitent maintenant explorer les possibilités de trouver des molécules inhibitrices capables de se fixer sur les différentes conformations identifiées. Ces inhibiteurs, bloquant la fonction de l'enzyme NadA, pourraient alors être éligibles pour la conception de nouveaux antibiotiques.
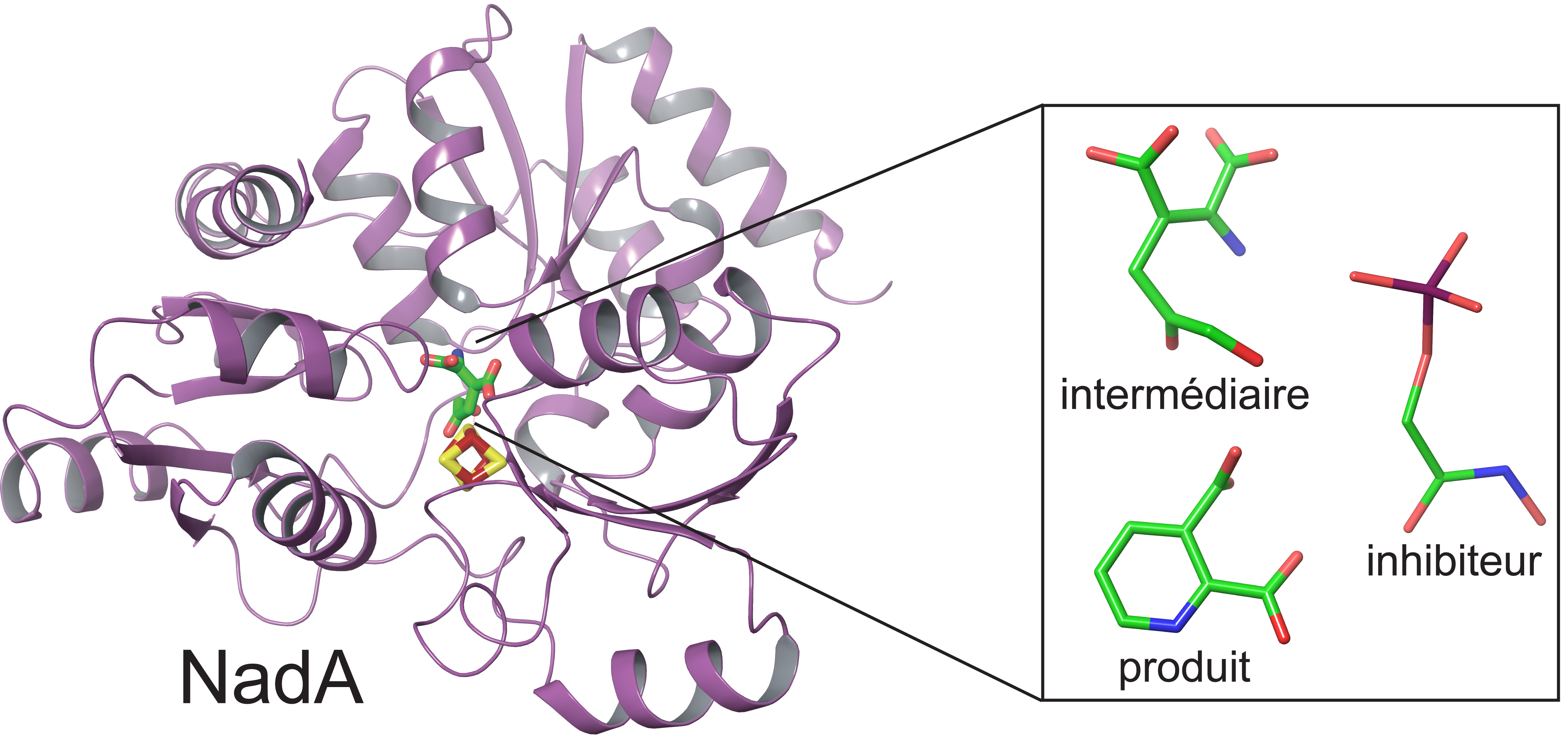 |
| Structure en rubans de la quinolinate synthétase (NadA, en mauve) avec son centre fer-souffre (en rouge et jaune au centre de la molécule). A droite, on distingue les 3 molécules fixées dans les cristaux de NadA. Ces molécules ont permis de définir le mécanisme catalytique de cette enzyme. |