La cryo-microscopie électronique (cryo-ME) est une technique qui permet d'obtenir la structure 3D d'échantillons biologiques avec une précision quasi-atomique. Depuis quelques années et les avancées de l'Intelligence Artificielle, elle est complétée de logiciels qui utilisent des algorithmes de prédiction et de construction de modèles atomiques de protéines.
En utilisant ces approches complémentaires, des chercheurs de l'institut de biologie structurale (CEA-Irig), de l'ESRF et de l'EMBL Grenoble ont révélé pour la première fois, à une échelle quasi-atomique, la structure complète de la nucléocapside (ensemble formé par le génome viral et son enveloppe protectrice appelée capside) d'un Baculovirus.
Compléter les données de la cryo-ME avec les logiciels Modelangelo et Alphafold
Les Baculovirus sont des virus qui infectent les insectes. Ils sont fréquemment utilisés en agronomie mais aussi dans les biotechnologies, notamment pour produire des protéines recombinantes. Malgré cette utilisation courante dans des domaines stratégiques, les connaissances à l'échelle moléculaire de l'assemblage de la nucléocapside de ce virus restent limitées. Avant cette étude, de nombreuses protéines structurales n'étaient pas encore localisées ni même identifiées.
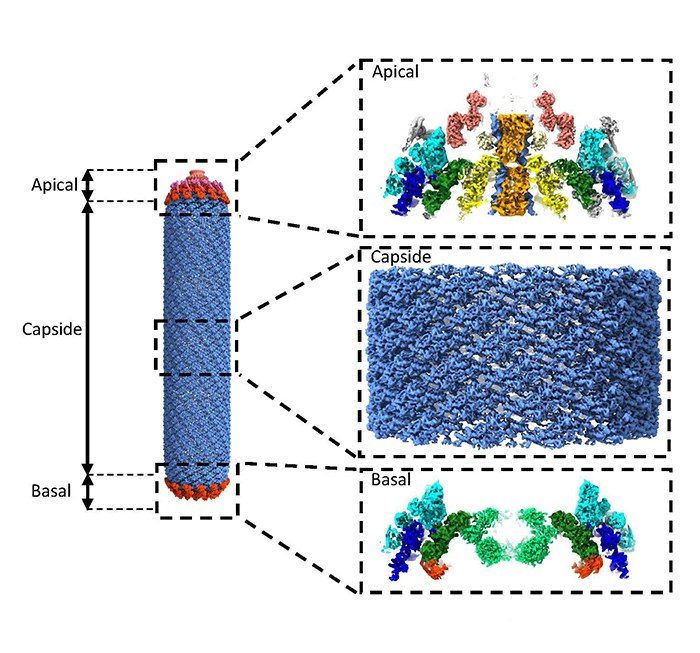
Figure : Structure de la nucléocapside entière obtenue par cryo-ME. A droite, de haut en bas : zoom sur une vue de côté du capuchon « apical » (la « porte d'entrée et de sortie » de l'ADN viral), la capside, une vue de côté de la structure « basale ». © CEA-Irig/IBS/G. Effantin
La nucléocapside forme une structure allongée de 50 nm de large et d'environ 300 nm de long en moyenne dans laquelle on distingue deux parties terminales distinctes : le capuchon « apical » et la structure « basale » (figure ci-dessus). Les chercheurs se sont particulièrement intéressés à ces deux structures.
Avec la cryo-ME, ils ont d'abord pu élucider différentes symétries dans la nucléocapside définissant ainsi plusieurs sous-ensembles protéiques dont ils ont déterminé les cartes 3D dans le but d'identifier et positionner les protéines les constituants. Ils ont ensuite travaillé ces données avec deux programmes d'apprentissage automatique :
- Modelangelo – conçu pour construire des modèles atomiques à partir de cartes de cryo-ME – a été utilisé pour identifier les constituants des cartes 3D ayant les plus hautes résolutions.
- Alphafold – conçu pour prédire la structure 3D des protéines à partir de séquences d'acides aminés – a été utilisé pour identifier les constituants des cartes 3D ayant de plus basses résolutions ainsi que pour suggérer d'éventuels partenaires d'interaction.
Cette approche a conduit à l'identification de huit protéines inédites et d'un segment d'ADN localisé dans le domaine apical, suggérant son rôle dans l'entrée et la sortie du génome. L'étude livre ainsi la première structure intégrale des parties symétriques de la nucléocapside et ouvre la voie à des outils biotechnologiques plus rationnels.